产品简介
结合三代HiFi和Ultra-long 等测序技术对基因组进行组装,获得高质量的基因组,尤其在端粒、着丝粒等位置有较好的组装结果。在组装结果中,至少有几条染色体是不包含gap的,整个基因组包含的gap数量也尽可能的少。
对于特定遗传元件研究,如端粒、着丝粒、重复元件和核糖体基因簇,以及结构重排、基因表达调控和核型进化等,高质量的基因组起到非常关键作用。
技术流程
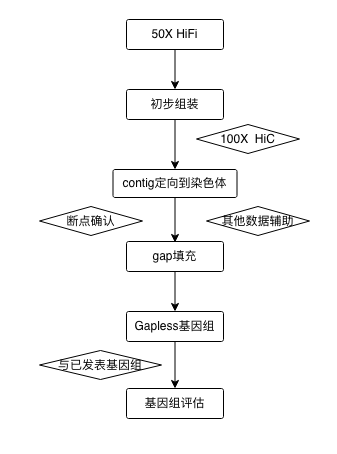
基因组测序策略
|
分析内容 |
文库类型 |
测序深度 |
|
基因组评估 |
DNA小片段文库 |
50X |
|
组装 |
HiFi文库 |
50X |
|
Ultra-long文库 |
100X |
|
|
HiC文库 |
100X |
|
|
注释 |
真核转录组文库 |
6G/样(5-6个不同组织) |
|
三代全长转录组文库 |
20-30G |
基因组样品准备
|
样本类型 |
DNA小片段文库 |
HiFi文库 |
Hi-C-文库 |
真核转录组文库 |
三代全长转录组文库 |
|
植物 组织 |
≥0.5g |
≥4g |
≥3g |
≥0.5g |
≥2g |
|
动物 组织 |
≥0.3g |
≥3g |
≥3g |
≥0.3g |
≥1.5g |
|
DNA (合格样品) |
≥0.5ug |
≥10ug(单cell) |
—— |
≥0.8ug |
≥1.6ug |
注意事项:
1) 各文库测序样本需保证来自同一植株或同一无性系;
2) 植物样本优先选取新鲜幼嫩叶片,对于根、茎等部位及多糖多酚类植物建议增加一倍送样量;动物样本优先选择肝脏、肾脏、肌肉等核酸含量较多组织;昆虫样本为去除腹部后的样本量;较小个体混样需保证亲本尽量一致;较大个体建议蛹期,非蛹期需饥饿处理,减少外源污染。
3) 三代全长转录组辅助注释混样建议不超过6个;
结果展示
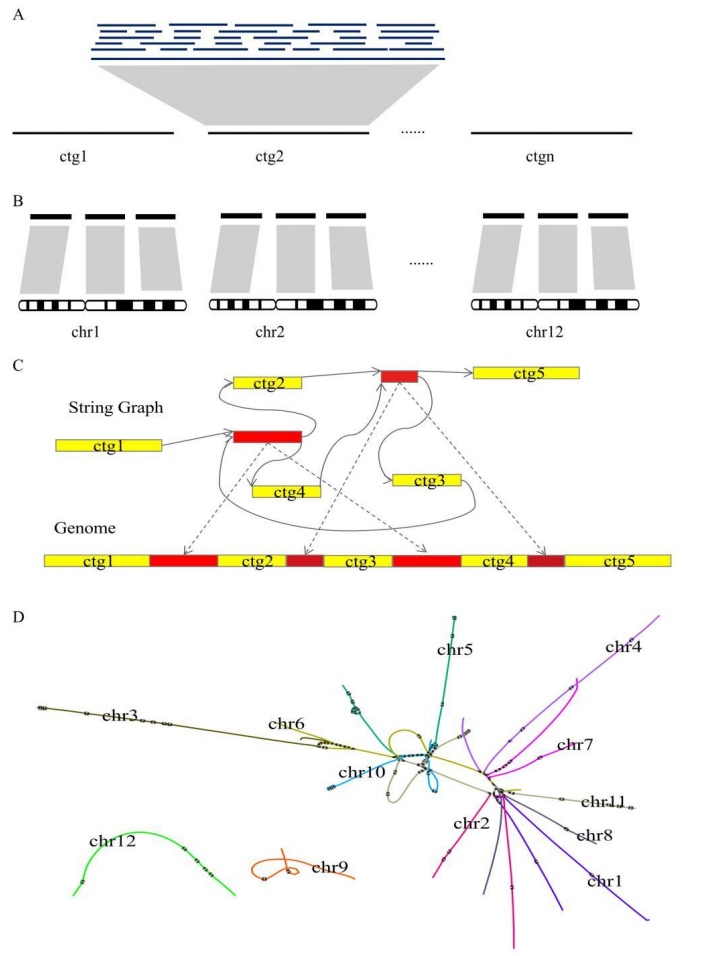
Gapless基因组组装示意图
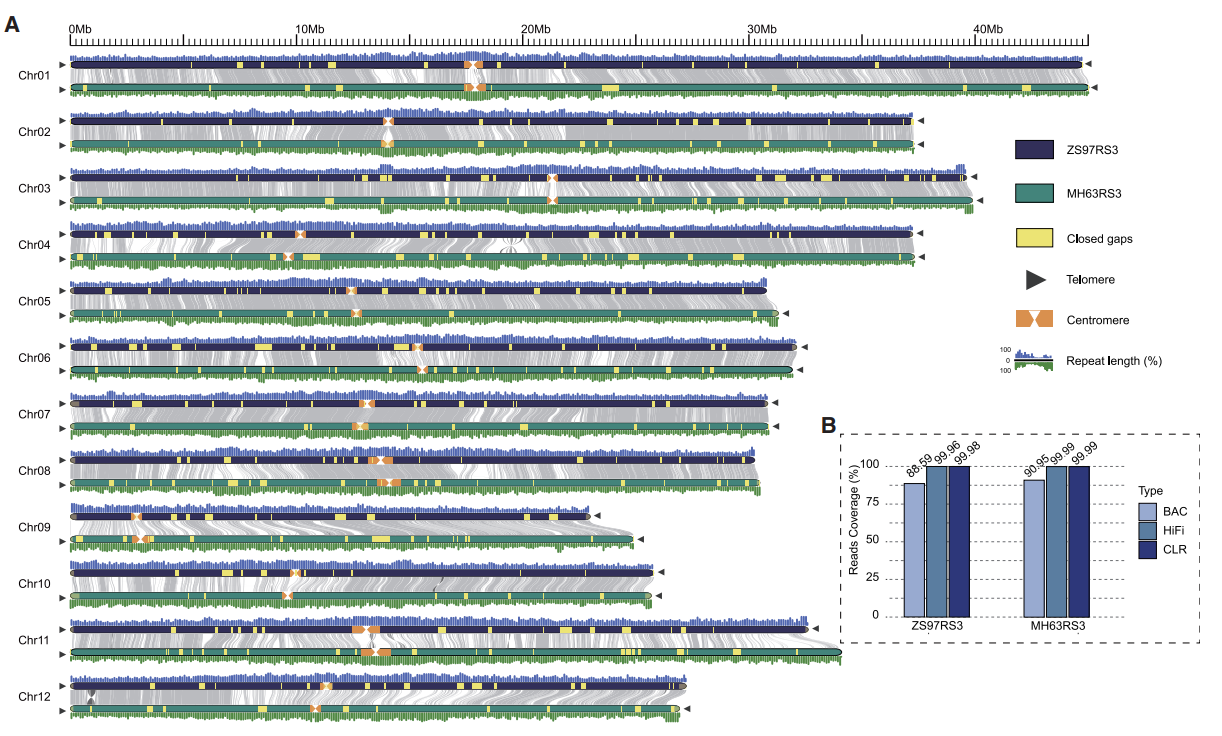
两个gap-free基因组
经典案例
|
物种 |
测序技术 |
发表期刊 |
年份 |
生物学问题 |
|
人[1] |
HiFi+ONT超长+Hi-C+Bionano |
Science |
2022年 |
获得了人类完整的基因组图谱,包括较难组装的区域即所有的着丝粒卫星阵列、近端重复区域、和5个端中心染色体的短臂,为后续遗传变异和功能研究奠定基础。 |
|
大刺鳅[2] |
HiFi+Hi-C |
Genome Biology |
2021年 |
通过基因组组装获得大刺鳅Y染色体完整序列,并提出了其性染色体近着丝粒的异染色质区域起源假说。 |
|
水稻[3] |
HiFi+基因组比对 |
Molecular Plant |
2021年 |
完成了2个gap-free水稻参考基因组,分析了水稻着丝粒结构,并对11号染色体末端水稻抗性相关的结构变异区域深入研究。 |
|
拟南芥[4] |
HiFi+ONT超长+Bionano |
Science |
2021年 |
组装了拟南芥5 条染色体含着丝粒全序列的基因组,并研究了着丝粒遗传进化和表观遗传特征。 |
参考文献
[1] Nurk Sergey, Koren Sergey, Rhie Arang, et al. The complete sequence of a human genome.[J]. Science (New York, N.Y.),2022,376(6588).
[2] Xue Lingzhan, Gao Yu, Wu Meiying, et al. Telomere-to-telomere assembly of a fish Y chromosome reveals the origin of a young sex chromosome pair.[J]. Genome biology,2021,22(1).
[3] Song JiaMing, Xie WenZhao, Wang Shuo, Guo YiXiong, et al. Two Gap-free Reference Genomes and a Global View of the Centromere Architecture in Rice.[J]. Molecular plant,2021,14(10).
[4] Naish Matthew, Alonge Michael, Wlodzimierz Piotr, et al. The genetic and epigenetic landscape of the Arabidopsis centromeres.[J]. Science (New York, N.Y.),2021,374(6569).

